L’Académie suédoise a récompensé les deux chercheuses pour la mise au point du système universel d’édition du génome Crispr-Cas9.
L’Académie suédoise a récompensé les deux chercheuses pour la mise au point du système universel d’édition du génome Crispr-Cas9.
https://www.nobelprize.org/prizes/chemistry/2020/summary/
Par Nathaniel Herzberg Publié aujourd’hui à 12h19, mis à jour à 12h49
http://https://www.lemonde.fr/sciences/article/2020/10/07/le-prix-nobel-de-chimie-a-la-francaise-emmanuelle-charpentier-et-l-americaine-jennifer-doudna-pour-les-ciseaux-moleculaires_6055125_1650684.html

Depuis quatre ans, on leur promettait le Nobel de médecine. La vénérable académie suédoise a fini par entendre les arguments… C’est dans la catégorie « chimie » qu’elle a attribué son prix, mercredi 7 octobre, à la Française Emmanuelle Charpentier et à l’Américaine Jennifer Doudna, pour leur découverte d’un outil moléculaire qui permet « de réécrire le code de la vie ».
Après les prix accordés en 2009 à l’Australienne Elizabeth Blackburn et l’Américaine Carol Greider, c’est la deuxième fois, seulement, depuis la création de la récompense, en 1901, que deux femmes sont simultanément honorées dans une même discipline.
Personne ne trouvera à redire à cette reconnaissance. La mise au point, en 2012, de ce que l’on a coutume de nommer « le couteau suisse de l’édition du génome », a sans aucun doute marqué une véritable révolution : en recherche médicale et en biochimie, mais bien au-delà. Avec Crispr, la balbutiante ingénierie du génome a été mise à la portée de tous les laboratoires. Des chercheurs en biologie fondamentale aux inventeurs des végétaux ou des animaux de demain, des médecins en quête de solution pour vaincre le paludisme ou faciliter les transplantations d’organes, à ceux qui traquent les maladies génétiques, tous ne jurent désormais que par Crispr-Cas9.
Le terrain foulé par les lauréates n’était certes pas vierge. Au milieu des années 1960, des scientifiques avaient découvert que certaines enzymes (dites « de restriction ») pouvaient couper l’ADN de certaines cellules à certains endroits… Mais l’affaire restait assez hasardeuse.
Au début du XXIe siècle, un premier bond a été entrepris : les « nucléases à doigt de zinc » et les « TALENs » ont permis de couper le génome à peu près là où on le souhaitait. Mais au prix d’une mise au point particulièrement lourde : chaque gène visé imposait en effet la construction d’une protéine spécifique. Crispr-cas9 a permis de passer du sur-mesure au prêt-à-porter sans rien perdre en précision. Simple, rapide, et donc bon marché, le système a conquis toute la sphère scientifique.
Son secret : la fameuse protéine Cas9. Capable de couper presque n’importe quoi n’importe où, elle doit juste être correctement guidée. Pour cela, il suffit de lui adjoindre une information génétique précise : la portion d’ARN correspondant à l’ADN visé. Au lieu de construire une protéine en trois dimensions, on compose une succession de paires de bases, autrement dit un problème à… une dimension. Ce qui prenait un an prend désormais moins d’une semaine et fonctionne chez les bactéries et les plantes, comme chez les souris et les hommes.
Une longue chaîne de chercheurs
Si, comme le prévoit son règlement, l’Académie a limité son palmarès à deux noms, c’est en réalité une longue chaîne de chercheurs qui a permis cette percée scientifique. Une histoire exemplaire liant la recherche la plus fondamentale et solitaire à la quête de solutions industrielles. L’Espagnol Francis Mojica, de l’université d’Alicante, en Espagne, a sans doute posé la première pierre à l’édifice. Passionné par le génome d’un organisme unicellulaire particulièrement résistant aux hautes concentrations salines dans les marais voisins, il y découvre de curieuses répétitions.
Elargissant sa recherche, il met en évidence la même structure dans une quarantaine de bactéries et archées. Avec un collègue hollandais, il leur donnera un nom : Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR), ce qui pourrait se traduire par « groupement d’éléments palindromiques courts répétés et régulièrement espacés ». En clair, des séquences identiques se répètent régulièrement dans le génome.
Surtout, Mojica retrouve, entre ces séquences, des morceaux de génome… de virus, les pires ennemis des bactéries. Il émet donc l’hypothèse, en février 2005, que les Crispr constitueraient un système immunitaire : lors d’une première attaque, les bactéries survivantes intégreraient dans leur génome un bout de gène caractéristique de leur assaillant, ce qui leur permettraient de se défendre lors d’une attaque ultérieure.
Cette belle intuition, c’est une équipe française, installée à Dangé-Saint-Romain, dans la Vienne, qui va la vérifier expérimentalement. Les chercheurs de Danisco – racheté depuis par le géant de l’agrochimie DuPont – ont une cible : les phages, ces virus qui attaquent les bactéries du yaourt et interrompent le processus de fermentation. Après deux ans de minutieuse recherche, Philippe Horvath, Rodolphe Barrangou et leur collègue canadien Sylvain Moineau, apportent la preuve expérimentale de cette nouvelle immunité.
Le coup d’envoi d’une course mondiale
Publiée dans Science, la découverte lance le coup d’envoi d’une course mondiale. En Europe comme aux Etats-Unis, de nombreuses équipes s’attachent à détailler la structure et la biochimie du système Crispr-Cas9. Comment il enregistre l’identité des virus assaillants, comment il les intègre dans le génome de la bactérie, puis comment, lors d’attaques ultérieures, il part en chasse, vise, reconnaît et élimine les ennemis.
En juin 2012, les équipes d’Emmanuelle Charpentier – alors en poste à UMEA, en Suède – et Jennifer Doudna, de l’université de Berkeley, en Californie, parviennent à reconstituer l’intégralité du puzzle. Surtout, elles reproduisent le système in vitro, hors, donc, de sa bactérie initiale. Crispr peut désormais être programmé afin de viser n’importe quel gène, retrancher une séquence d’ADN, mais aussi la remplacer par une autre.
La porte est ouverte. La planète scientifique s’engouffre sur ces terres nouvelles et court après les applications. Cette fois, c’est sur la côte Est, à Boston, que l’Américain Feng Zhang (Broad Institute) réussit une nouvelle prouesse : il parvient, grâce à Crispr, à modifier le génome de cellules animales et humaines – des cellules dites « eucaryotes », c’est-à-dire possédant un noyau. Tout le vivant s’ouvre à présent aux lames des nouveaux « ciseaux moléculaires ».
Les uns en perfectionnent la précision, les autres en étendent les applications, d’autres encore, en mettent en évidence les limites. Des querelles sur les brevets sont toujours pendantes. Loin des laboratoires, juristes, philosophes et même responsables politiques tentent de leur côté d’en mesurer la portée, de la fabrication des « nouveaux OGM » à la quête du bébé parfait. Pour le pire ou le meilleur, une révolution est en marche. C’est aussi cela que le Nobel de chimie a reconnu.
Les articles de notre série « La saga Crispr-Cas9 »
Le premier épisode vous fait découvrir ce nouvel outil moléculaire, Crispr, qui permet de modifier les génomes à volonté, chez tous les êtres vivants, bouleversant les molécules.
Dans le deuxième épisode, apprenez comment, au milieu des années 2000, une découverte va révolutionner la biologie : les bactéries qui servent de ferment dans vos yaourts s’avèrent être une arme fatale contre les virus.
Troisième épisode. Amateurs de sciences, vous ne pouvez les avoir ratées : les biologistes française et américaine Emmanuelle Charpentier et Jennifer Doudna accumulent les prix pour avoir inventé les ciseaux moléculaires.
Dans ce quatrième épisode, découvrez comment Crispr-Cas9 est à l’origine de batailles sanglantes entre instituts rivaux et start-up.
Cinquième épisode. Leurs grands yeux écarquillés ont fait le tour du monde : début 2014, la revue « Cell » dévoile la naissance des deux premiers primates dont le génome a été modifié grâce à Cripsr-Cas9.
Dans le dernier épisode de cette série, découvrez comment en 2015 une expérience chinoise a été menée sur des embryons humains grâce à cet outil d’édition du génome.
Et aussi nos entretiens avec des acteurs majeurs de cette découverte :
Le généticien américain George Church évoque les nouvelles possibilités d’ingénierie du génome et son souhait de pouvoir les appliquer aux cellules germinales pour prévenir certaines maladies.
Le jeune chercheur américain Feng Zhang évoque les voies d’amélioration de ce système révolutionnaire d’édition génétique.
Le directeur du Broad Institute (MIT et Harvard), Eric Lander, revient sur le processus de découverte du système d’édition génétique Crispr, qu’il a récemment décrit dans un long récit, qui a suscité de nombreuses critiques.
Pour la biologiste américaine Jennifer Doudna, codécouvreuse du système d’édition du génome, les questions éthiques doivent être rendues publiques.
Spécialiste en biotechnologie, Rob Carlson estime que les politiciens et les industriels devraient être responsables de la façon dont ils utilisent la science.









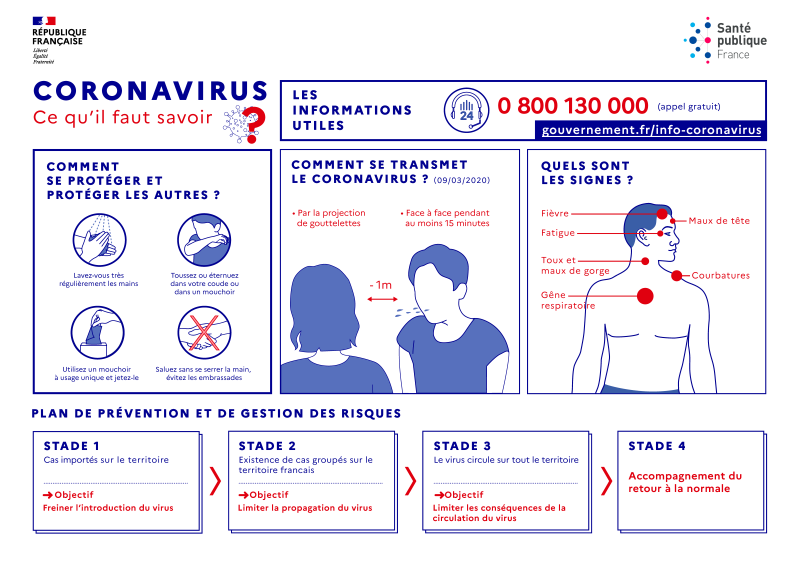
 Au cours des huit dernières semaines, l’Établissement français du sang est parvenu, grâce à la formidable mobilisation des donneurs, à répondre à tous les besoins de transfusion des patients.
Au cours des huit dernières semaines, l’Établissement français du sang est parvenu, grâce à la formidable mobilisation des donneurs, à répondre à tous les besoins de transfusion des patients.